Dihydrolipoyl déshydrogénase
| Dihydrolipoyl déshydrogénase | |||||||||||||||||||
 Structure d'une E3 humaine (PDB 1ZY8[1]) | |||||||||||||||||||
| Caractéristiques générales | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Symbole | DLD | ||||||||||||||||||
| N° EC | 1.8.1.4 | ||||||||||||||||||
| Homo sapiens | |||||||||||||||||||
| Locus | 7q31.1 | ||||||||||||||||||
| Masse moléculaire | 54 177 Da[2] | ||||||||||||||||||
| Nombre de résidus | 509 acides aminés[2] | ||||||||||||||||||
| Liens accessibles depuis GeneCards et HUGO. | |||||||||||||||||||
| |||||||||||||||||||
La dihydrolipoyl déshydrogénase (DLD), également appelée dihydrolipoamide déshydrogénase, est la dernière des trois enzymes du complexe pyruvate déshydrogénase (PDC), du complexe alpha-cétoglutarate déshydrogénase et du complexe 3-méthyl-2-oxobutanoate déshydrogénase, constitués chacun d'une décarboxylase, d'une transacétylase et d'une réductase.
Le complexe pyruvate déshydrogénase
Le PDC catalyse la décarboxylation oxydative du pyruvate en acétyl-CoA, réaction qui réalise notamment la liaison entre la glycolyse et le cycle de Krebs ; les autres enzymes du complexe sont la pyruvate déshydrogénase (E1) et la dihydrolipoamide S-acétyltransférase (E2). Cette réaction s'écrit globalement :
 | + CoA-SH + NAD+ → NADH + H+ + CO2 + |  |
| Pyruvate | Acétyl-CoA |
Le mécanisme en est assez complexe, et peut être résumé par le schéma simplifié ci-dessous :

- la pyruvate déshydrogénase (E1) catalyse les étapes A et B avec la thiamine pyrophosphate (TPP),
- la dihydrolipoamide S-acétyltransférase (E2) catalyse l'étape C avec le lipoamide et la coenzyme A (CoA-SH),
- la dihydrolipoyl déshydrogénase (E3) catalyse les étapes D et E avec la FAD et la NAD+.
La dihydrolipoyl déshydrogénase
| N° EC | EC 1.8.1.4 |
|---|---|
| N° CAS | 9001-18-7 |
| Cofacteur(s) | FAD |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
modifier 
La DLD est une flavoprotéine qui, outre sa participation aux trois complexes enzymatiques que sont le complexe pyruvate déshydrogénase, le complexe alpha-cétoglutarate déshydrogénase et le complexe 3-méthyl-2-oxobutanoate déshydrogénase, catalyse plus simplement la réduction du lipoamide en dihydrolipoamide.
Son confacteur est la flavine adénine dinucléotide (FAD), qui transfère au nicotinamide adénine dinucléotide (NAD+) les deux atomes d'hydrogène libérés par le dihydrolipoamide lors de son oxydation en lipoamide.
 | 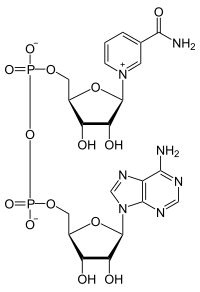 | |
| Flavine adénine dinucléotide (FAD) | Nicotinamide adénine dinucléotide (NAD+) |
Notes et références
- ↑ (en) Ewa M. Ciszak, Anna Makal, Young S. Hong, Ananthalakshmy K. Vettaikkorumakankauv¶ Lioubov G. Korotchkina et Mulchand S. Patel, « How Dihydrolipoamide Dehydrogenase-binding Protein Binds Dihydrolipoamide Dehydrogenase in the Human Pyruvate Dehydrogenase Complex », Journal of Biochemical Chemistry, vol. 281, no 1, , p. 648-655 (PMID 016263718, DOI 10.1074/jbc.M507850200, lire en ligne)
- ↑ a et b Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
 Portail de la biochimie
Portail de la biochimie










